Imagestudio V3
Imagestudio V3
交互分析中的图像分析是时空组学图像处理软件,主要涉及图像质量评估和图像手动调整模块(图像拼接、图像校准、组织分割和细胞分割)。图像质量评估主要从图像清晰度和图像track线两个评估指标对显微镜拍摄的图像进行评分,用于判断是否满足下游的生信分析。图像的手动调整模块,主要针对自动的拼接、校准或者分割算法无法满足下游进一步分析的需要时,借助手动工具对图像进行处理。
图像拼接(小图场景)
图像拼接模块用于手动调整显微镜小图的拼接效果。
注意:输入文件应为小图的场景,大图场景暂不适用。
基本操作
- 在云平台进入某个项目中。

- 点击“交互分析”的“图像分析”。

- 点击“图像拼接”


- 选择待手动拼接的文件(IPR)界面。

用户可根据文件名称、SN 号和任务 ID 搜索对应的 IPR 。
打开文件支持的类型为:IPR
输出文件的存储路径为: 数据管理->ManualData->Stitch

- 点击“确认”,选择拼接方法,根据数据展示适配的拼接方法并用黄色高亮。

- 若是选择的“track线拼接”,系统会基于打开的IPR文件(记录QC模块检测出的每张小图FOV的track线交叉点信息),生成一张局部大图。界面中的每个小方块展示每个FOV小图中track线交点的局部图,并根据小图行列号拼成局部大图。

说明:若是DAPI&mIF的数据,系统会基于DAPI生成一张局部大图。
点击“不再提示”,若是不关闭软件的情况下这个FOV说明将不再展示。
点击“我知道了”,关闭当前的提示。若是想再次打开,可点击“视图”的“FOV说明”。
若是选择的“无track线拼接”
- 系统会基于打开的IPR文件(记录QC模块检测出的每张小图FOV的track线交叉点信息)以及小图TAR.GZ文件,生成一张拼接后的全局大图。界面中的每个小方块代表每个FOV,并根据小图行列号拼成一张全局大图。


- 拖动“拼接误差调节”,全局大图中将展示算法检测到大于该误差的FOV并标记为红色。设置的误差越小,全局大图中红色的FOV越多。

Track线模板的手动调节
局部大图中标记为M的FOV为模版FOV,通常为track线QC评估最优的FOV。ImageStudio通过算法根据模版FOV识别的track线推导出全图的track线情况,可检查模版FOV识别出的track线(绿色)是否与底图图像的track线重合,预估拼接效果。
每个类型代表的含义如下:
X: QC未检测到的track点,需用户协助是否可肉眼标记track点。
M: QC找到的最佳模板点,需用户协助算法标记的网格线效果是否良好。
○: 算法评估的拼接误差小于5个像素,用户可协助确认效果。
○: 算法评估的拼接误差大于5个像素,需用户协助肉眼标记track点。
建议用户操作:
首先检查M点标记是否正确,若是不正确,需要手动调整M(必须)。
其次检查红色圆圈标记的FOV,该FOV算法评估拼接误差在5像素以上(必须)。
再次检查红色X的FOV,该FOV是算法找不到track线的,需要看下肉眼是否可以看到并标记(可选)。
最后可协助检查绿色圆圈的FOV即可(可选)。
M标记的FOV
主要调节track网格线,尽量将算法生成的绿线跟芯片的track线对齐。
- 双击局部大图标记M的FOV或者单击左侧FOV的列表打开。

- 调整滑块增强图像,并按下Ctrl+鼠标滚轮放大后确认推导模版线和底图track线是否对齐。

- 如果没有对齐,点击“M”图标修改 track 网格线。

- 出现红色十字线和黄色网格线。红色线为选中聚焦的track线,旋转缩放等操作均以此track线为中心操作。黄色线为非聚焦的网格线。

- 找到清晰的track点,点击鼠标左键即可将聚焦的红色track线移至点击的图像track线交点处。

- 缩小图片,可通过“模板调节”面板的上下左右键,在水平和垂直方向调整黄线,确保和图片track对齐。此操作最多不超过9次。

- 按下鼠标左键点击“模板调节”的“角度调节”以红色聚焦track线为中心调整旋转角度。

- 按下鼠标左键点击“模板调节”的“尺度调节”的加号和减号,以红色聚焦track线为中心调整旋转角度。减号为缩小尺度,加号为放大尺度。
非模板FOV调节
主要调节标记红色X和红色○的点。
- 双击带有红色X或者红色o的小图FOV,进入调节非模板FOV的位置。


- 调节图像增强,找到一个清晰的十字track点,点击“+”图标,图上会出现一个绿色圆圈,鼠标移动到该点上,出现小手状态,可拖动此点移至实际track线交点位置,移动过程中绿色圆圈会变为红色十字线,以便于与track线比较。



- 若对于位置不满意,选中该点,鼠标左键直接将绿色圆圈拖动到任何位置。

- 编辑完成,默认编辑后FOV的状态都是已确认。若是对某个FOV位置不确认,可设置状态为待检查。

- 修改后,点击“ssDNA页签”或者“Home”,返回局部拼接大图的页面。非模板FOV在局部拼接大图中已确认和待核查状态分别标记为用绿色圆圈+√ 和黄色感叹号。已确认的表示track交点已确认调整完毕,待核查的表示track交点位置需要重新核查。

- 全图调整完毕后点击“运行”,若是存在待核查FOV,系统会提醒是否检查后运行。

- 若是全部为已确认的FOV,点击“运行”程序会根据手动标记的局部拼接大图中的每个手动选中的track交点坐标点的位置重新拼接。

- 运行完成会基于当前的手动拼接效果给出评分,并提示是否打开拼接好的大图。

- 若是“确认”打开,会将拼接大图推导的track线打在拼接大图上,可确认track线是否与芯片上的线对齐。

图像重叠区域的手动调节
基于图像重叠区域的大图中标记为红色圆圈的FOV,通常为自动算法评估该FOV的拼接误差大于设定的值(默认值5个像素),可以拖动右侧的“拼接误差调节”按钮,红色圆圈的个数会随着误差的减小逐渐增多。
- 双击全局大图中红色的FOV或者单击左侧FOV的列表打开。

- 生成一张九宫格的小图,调节图像增强后,可在右侧“拼接调节”中移动圆盘或者键盘的上下左右方向键调节FOV的位置。

- 相邻FOV的拼接效果可以根据OverLap的分数或者颜色的重合程度(黄色)来判断。
 注意:OverLap的分数区间是0~100之间,分数越高说明相邻FOV的重合度越高,拼接误差相对较小。
注意:OverLap的分数区间是0~100之间,分数越高说明相邻FOV的重合度越高,拼接误差相对较小。
- 调整完成后,“保存”,点击“ssDNA”或者“Home”页签,返回全局大图的页面,修改后的FOV在全局大图中用“对钩”标识。

- 全图FOV调整完毕后点击“运行”,程序会根据手动移动的FOV重新拼接新的大图。

- 运行完成后,提示是否打开拼接好的大图。

- 点击“确认”,将打开拼接后的大图可以查看拼接的效果。

组织分割
组织分割模块用于手动调整分割后的细胞Mask。
基本操作
- 在云平台进入某个项目中。

- 点击“交互分析”的“图像分析”。

- 点击“组织分割”


- 选择待手动分割的文件(IPR)界面。

用户可根据文件名称、SN 号和任务 ID 搜索对应的 IPR 。
打开文件支持的类型为:IPR
输出文件的存储路径为: 数据管理->ManualData->Image->TissueSeg

- 若是"ssDNA/DAPI",点击“确认”,系统基于打开的文件生成底图或者组织Mask,是否生成组织Mask页面取决于用户输入的IPR文件中是否做过组织分割。

若是DAPI&mIF,点击“确认”。
系统基于打开的文件生成DAPI底图或者组织Mask页面,是否生成组织Mask页面取决于用户输入的IPR文件中是否做过组织分割。

- 用户可在Tab页签勾选要打开其他蛋白底图和对应的Mask页面,比如选择TESK2。

- 打开TESK2后的界面如下。

- 用户可在Tab页签取消勾选要打开其他蛋白底图和对应的Mask页面。

- 点击“取消”,返回前一个页面。

注意:因打开文件会生成金字塔比较耗时,当前只支持打开一张芯片数据。1c****m*1cm芯片T时间大概在3min~5min。
矩形
主要基于原图页面,根据框选的区域,运行组织分割算法,并将组织Mask并写入IPR文件中。
- 在原图页面,点击“矩形”。

- 选择是“替换Mask”或者“更新Mask”,默认是替换Mask。

说明:
“替换Mask”主要是根据框选区域生成的Mask,替换打开的Tissue_Mask。
“更新Mask”主要是根据框选区域生成的Mask,更新框选区域生成的Tissue_Mask,其他区域的Tissue_Mask不变。
- 框选感兴趣的区域。

- 点击“运行”。

- 算法会基于框选区域,提取组织Mask文件并写入IPR文件中。

- 若输入的数据未做过组织分割,矩形运行完,则会生成新的“Mask”页签。



多边形
主要基于原图页面,手动画Mask组织轮廓,将框选区域与原来的Mask做叠加并写入IPR文件中,支持一次画多个。
- 在 Mask 页面,点击“多边形”。

- 从一个点起步,点击鼠标左键增加点,最终设置成封闭的多边形。

说明:当前不支持重叠区域绘制。
- 按住键盘Enter,可结束多次标记,鼠标右键标注的多边形区域,进入多边形编辑状态。

- 点击多边形轨迹的点,按住鼠标左键可拖拽多边形的形状。

- 在保存之前可点击“撤销”按钮,撤销当前标注的操作。

- 画完之后点击“保存”,生成新的Mask并写入IPR文件。


- 点击“游标”可退出多边形编辑状态。
画笔
主要基于Mask页面,根据自动算法运行的Mask文件,手动画组织掩膜Mask。
- 在Mask页面,点击“画笔”。

- 点击画笔下面的“三角”符号,可调节画笔的大小。范围为[0,100],单位是像素。

- 调节“透明度”可查看未分割的组织轮廓区域,按住鼠标左键画轮廓。

注意:在画笔操作过程中,可点击右下方视野小地图拖动图片位置。
- 若画笔误画,在保存之前可点击“撤销”按钮,撤销当前操作。

- 画完之后点击“保存”,更新Mask并写入IPR文件。


- 点击“游标”,可退出画笔编辑状态。
橡皮擦
主要基于Mask页面,根据自动算法运行的Mask文件,手动擦除Mask组织掩膜。
- 在Mask页面,点击“橡皮擦””。

- 点击橡皮擦下面的“三角”符号,可调节橡皮擦的大小。范围为[0,200],单位是像素。

- 调节“透明度”可查看是否有过多分割的组织轮廓,按住鼠标左键可移动擦除。

注意:在橡皮擦操作过程中,可点击右下方视野小地图拖动图片位置。
- 若橡皮擦误擦,在保存之前可点击“撤销”按钮,撤销当前操作。
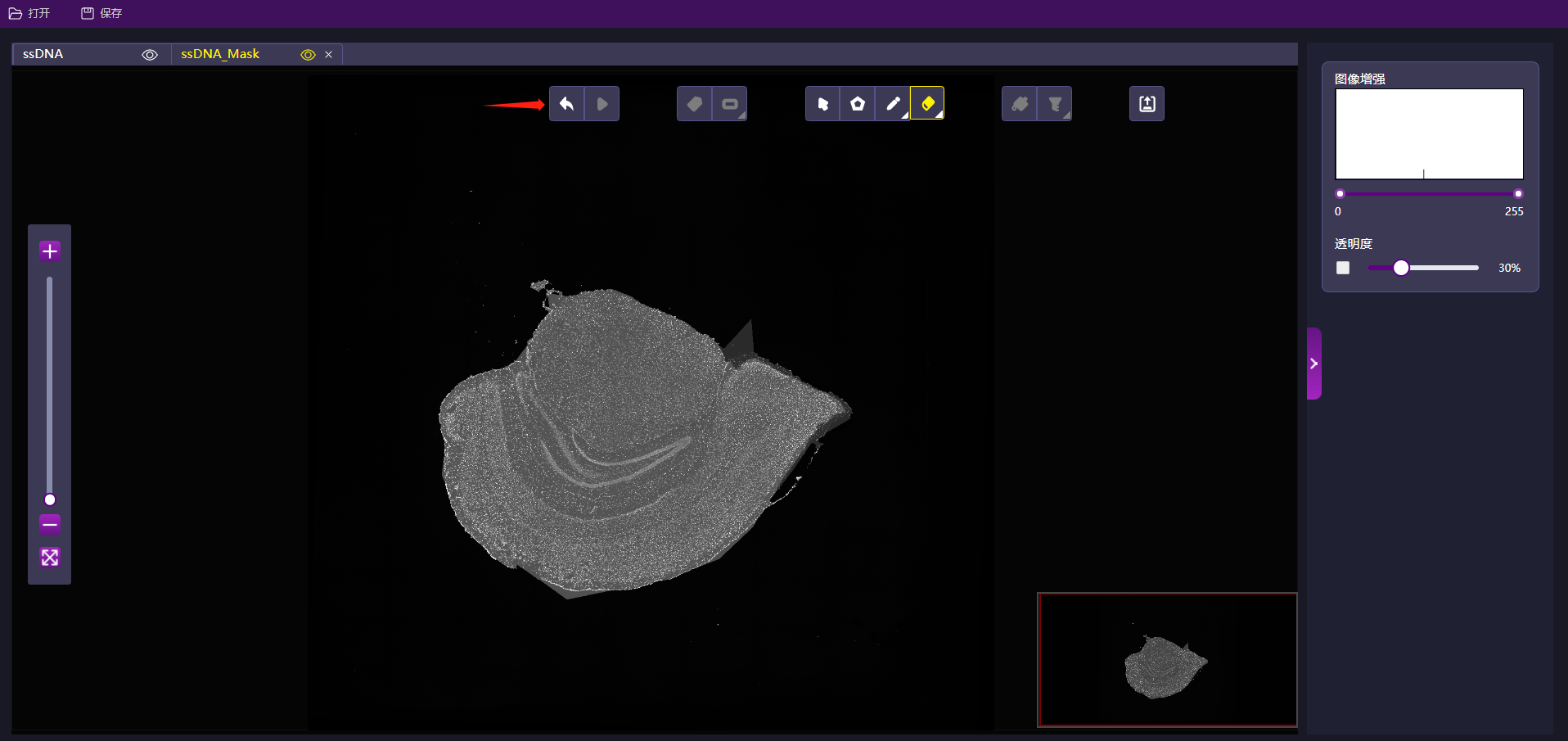
- 擦完之后点击“保存”,更新Mask并写入IPR文件。


- 点击“游标”,可退出橡皮擦编辑状态。
空洞填充
主要基于Mask页面,针对组织区域的空洞,框选填充区域做手动填充。
- 在Mask页面,点击“空洞填充”。

- 鼠标右键点击需要填充的空洞区域,算法将自动填充图像的空洞。

- 运行完空洞填充,更新Mask并写入IPR文件。

- 点击“游标”,可退出空洞填充编辑状态。
注意:空洞的定义为被白色Mask包围的黑色区域。
过滤器
主要基于Mask页面,过滤拍照过程中的图像噪声杂质。
- 在Mask页面,拖动过滤器的参数。

- 拖动过滤器参数,可在Mask页面查看过滤效果。

- 点击“保存”,更新后的Mask写入IPR文件。


- 可同时使用游标、画笔和橡皮擦。
阈值
主要基于DAPI&mIF,在蛋白的Mask页面,通过阈值调节蛋白Mask。
- 在蛋白Mask页面,调节“透明度”,将原图和Mask的重合程度更好的体现。

- 点击“眼睛”隐藏其他蛋白,避免其他蛋白的展示对当前蛋白的调节造成影响。

注意:每个图层的层级顺序从左到右依次增加,比如DAPI为最底层,其他蛋白依次类推。
- 在蛋白Mask页面,调节“阈值”。

注意:弹框提示,阈值更新会导致画笔和橡皮擦等操作不可见,重置或者保存后方可显示,建议用户先用阈值调节Mask,再用画笔、橡皮擦等其工具调节。
- 可点击“重置”按钮,恢复到系统默认值。

- 若操作有误,在保存之前可点击“撤销”按钮,撤销当前操作。

- 全部蛋白调节完成以后,点击“保存”,更新后的Mask写入IPR文件。

- 可同时使用游标、画笔和橡皮擦。
标注
主要基于H&E图像,对病例区域做注释,支持标注多个不同的组织区域,与组织分割标注整个区域是不同的。用户可以在HE_Label的tab页签中,使用画笔、橡皮擦或者多边形工具标注自己感兴趣的区域,比如癌区和正常区域。
说明:标注功能只适用于3.0.x及以上版本的H&E图像数据。
- 点击“标注”,生成HE_Label的Tab页签。

- 右侧点击菜单图标,选择“新建Label”,弹框填写Label的名字和备注信息,点击“确定”。


注意:Label的命名只能是数字、字母或者数字、字母、‘_’的组合。
- 可以使用多边形、画笔和橡皮擦工具,手动标记感兴趣的组织区域。

- 若操作有误,在保存之前可点击“撤销”按钮,撤销当前操作。

- 点击“保存”,在标注标签生成对应的Label,默认赋予不同的颜色并将更新后的Label_Mask写入IPR文件。

- 点击每个Label的眼睛,可以显示或者隐藏某个Label。或者在右侧的空白区域,选择“全部显示”或者“全部隐藏”,即可将显示/隐藏的Label全部关闭/打开。


- 如果对某个标注的Label不感兴趣,选中右侧列表的某个Label,鼠标右键,选择“删除Label”。

- 如果想编辑某个Label:
- 右侧选择“编辑Label”按钮,可以修改 label 的名字或者备注信息。 比如:修改为“Label_03” 。

- 点击“确认”,进入编辑Label的状态,提示按键盘的“ESC”退出编辑。

- 点击多边形、画笔和橡皮擦工具,手动标记感兴趣的组织区域,点击“保存”。

注意:为避免其他Label造成影响,编辑过程中可设置为隐藏其他Label。
细胞分割
细胞分割模块用于手动调整分割后的细胞Mask。
基本操作
- 在云平台进入某个项目中。

- 点击“交互分析”的“图像分析”。

- 点击“细胞分割”


- 选择待手动分割的文件(IPR)界面。

用户可根据文件名称、SN 号和任务 ID 搜索对应的 IPR 。
打开文件支持的类型为:IPR
输出文件的存储路径为: 数据管理->ManualData->CellSeg

- 若是"ssDNA/DAPI",点击“确认”,系统基于打开的文件生成底图或者组织Mask,是否生成组织Mask页面取决于用户输入的IPR文件中是否做过组织分割。

若是DAPI&mIF,点击“确认”。
- 系统基于打开的文件生成DAPI底图或者组织Mask页面,是否生成组织Mask页面取决于用户输入的IPR文件中是否做过组织分割。

- 用户可在Tab页签勾选要打开其他蛋白底图和对应的Mask页面,比如选择TESK2。

- 打开TESK2后的界面如下。

- 用户可在Tab页签取消勾选要打开其他蛋白底图和对应的Mask页面。

- 点击“取消”,返回前一个页面。

注意:因打开文件会生成金字塔比较耗时,当前只支持打开一张芯片数据。1c****m*1cm芯片T时间大概在3min~5min。
矩形
主要基于原图页面,根据框选的区域,运行细胞分割算法,并将细胞Mask写入IPR文件中。
- 在原图页面,点击“矩形”。

- 选择是“替换Mask”或者“更新Mask”,默认是更新Mask。

说明:
“替换Mask”主要是根据框选区域生成的Mask,替换打开的Cell_Mask。
“更新Mask”主要是根据框选区域生成的Mask,更新框选区域生成的Cell_Mask,其他区域的Cell_Mask不变。
- 框选感兴趣的区域。

- 点击“运行”。

- 算法会基于框选区域,生成新的Mask并写入IPR文件中。

- 若输入的数据未做过细胞分割,矩形运行完,则会生成新的“Mask”页签。


多边形
主要基于"ssDNA/DAPI"和Mask页面,手动画细胞轮廓,将框选区域与原来的Mask做叠加并写入IPR文件中,支持一次画多个。
- 在原图页面或者Mask页面,点击“多边形”。

- 选择需要手动标记的细胞,从一个点起步,点击鼠标左键增加点,最终设置成封闭的多边形。


- 按住键盘Enter,可结束多次标记,鼠标左键多边形区域,进入多边形编辑状态。

- 点击多边形轨迹的点,按住鼠标左键可在拖拽多边形的性状。

- 在保存之前可点击“撤销”按钮,撤销当前标注的操作。

- 画完之后点击“保存”,生成新的Mask并写入IPR文件。

- 点击“游标”可退出多边形编辑状态。
画笔
主要基于Mask页面,根据自动细胞分割算法运行的Mask文件,手动画Mask细胞掩膜。
- 在Mask页面,点击“画笔”。

- 点击画笔下面的“三角”符号,可调节画笔的大小。范围为[0,50],单位是像素。

- 调节“透明度”可查看未分割的细胞轮廓区域,按住鼠标左键画轮廓。

注意:在画笔操作过程中,可点击右下方视野小地图拖动图片位置。
- 若画笔误画,在保存之前可点击“撤销”按钮,撤销当前操作。

- 画完之后点击“保存”,更新Mask并写入IPR文件。


说明:更新后的数据会用红色填充展示,方便用户查看。
- 点击“游标”,可退出画笔编辑状态。
橡皮擦
主要基于Mask页面,根据自动分割算法运行的Mask文件,手动擦除Mask细胞掩膜。
- 在Mask页面,点击“橡皮擦”。

- 点击橡皮擦下面的“三角”符号,可调节橡皮擦的大小。范围为[0,100],单位是像素。

- 调节“透明度”可查看是否有过多分割的细胞轮廓,按住鼠标左键可移动擦除。

注意:在橡皮擦操作过程中,可点击右下方视野小地图拖动图片位置。
- 若橡皮擦误擦,在保存之前可点击“撤销”按钮,撤销当前操作。

- 擦完之后点击“保存”,更新Mask并写入IPR文件。


- 点击“游标”,可退出橡皮擦编辑状态。
标记
主要基于Mask页面,针对稀疏区域的细胞,通过点击细胞区域,利用算法快速识别细胞。
- 在Mask页面,点击“标记”。

说明:在原图页面可调节自动增强,达到组织区域和背景有较强的对比度,标记算法效果相对会比较好。
- 鼠标右键点击,提示获取细胞Mask,支持多次点击。

- 标记完点击“保存”,会将标记的细胞Mask写入IPR。

说明:因保存时间较久,建议全部标记完点击保存。
- 点击“游标”,可退出标记编辑状态。
图像校准
图像校准模块用于蛋白与DAPI的校准,手动调节蛋白图层,在形态学上尽量跟DAPI图层重合。支持在水平、垂直方向、小角度和小尺度的调节。
注意:输入文件为DAPI&mIF图像QC后的数据。
基本操作
- 在云平台进入某个项目中。

- 点击“交互分析”的“图像分析”。

- 点击“图像校准”


- 选择待手动校准的文件(IPR)界面。

用户可根据文件名称、SN 号和任务 ID 搜索对应的 IPR。
打开文件支持的类型为:IPR
输出文件的存储路径为: 数据管理->ManualData->Calibration

- 点击“确认”系统基于打开的文件生成DAPI和蛋白的页面。


注意:因打开文件会生成金字塔比较耗时,当前只支持打开一张芯片数据。1cm*1cm芯片T时间大概在3min~5min。
校准调节
- 打开文件,在蛋白页面可看到“校准调节”的面板。

- 在蛋白页面,调节“透明度”,将蛋白和DAPI的重合程度更好的体现。

- 点击“眼睛”隐藏其他蛋白,避免其他蛋白的展示对当前蛋白的调节造成影响。

在蛋白页面:
- 点击“校准调节”面板的中心可以设置调节的步长。

- 点击“校准调节”面板的上下左右键,在水平和垂直方向调整蛋白的位置。

- 点击"角度调节"和“尺度调节”,小角度和尺度调整蛋白图层的缩放和旋转角度。

注意:
用户可根据需要输入步长,默认“校准调节”的步长为1像素。
用户可点击键盘的上下左右键在水平和垂直方向移动蛋白的位置。
- 若移动有误,在保存之前可点击“撤销”按钮,撤销当前操作。

- 若撤销多次后,想重做上一个操作,可点击“重做”。

- 若部分蛋白未调节完成,点击“保存”,系统提示因保存花费时间较久,是否全部蛋白校准完成,才保存。

若选择“取消”系统返回保存前界面。
若选择“确认”更新后的信息写入IPR文件。
注意:因保存过程会生成图像金字塔,建议全部蛋白调节完成以后,在进行保存操作。
手动后数据路径说明
手动后的数据会存储在数据管理, 数据可同步到数据管理的三种条件。
手动操作完后,点击“保存”,执行如下两个操作数据会写入数据管理,用户可以投递后面的分析流程。
关闭页面5min 后,系统会将数据写入到数据管理。
若是 30min 内无新的操作,系统会自动将数据写入到数据管理。
点击各个模块的“投递流程”按钮,系统会将数据写入到数据管理并弹框填写参数的界面。
手动后写入数据管理的目录。
| 模块 | 数据管理的路径 |
|---|---|
| 图像拼接 | ManualData\Image\Stitch |
| 图像校准 | ManualData\Image\Calibration |
| 组织分割 | ManualData\Image\TissueSeg |
| 细胞分割 | ManualData\Image\CellSeg |
注意:因“保存数据”时间和内存消耗较大,建议用户将所有手动操作完后,点击“保存数据”。
流程投递说明
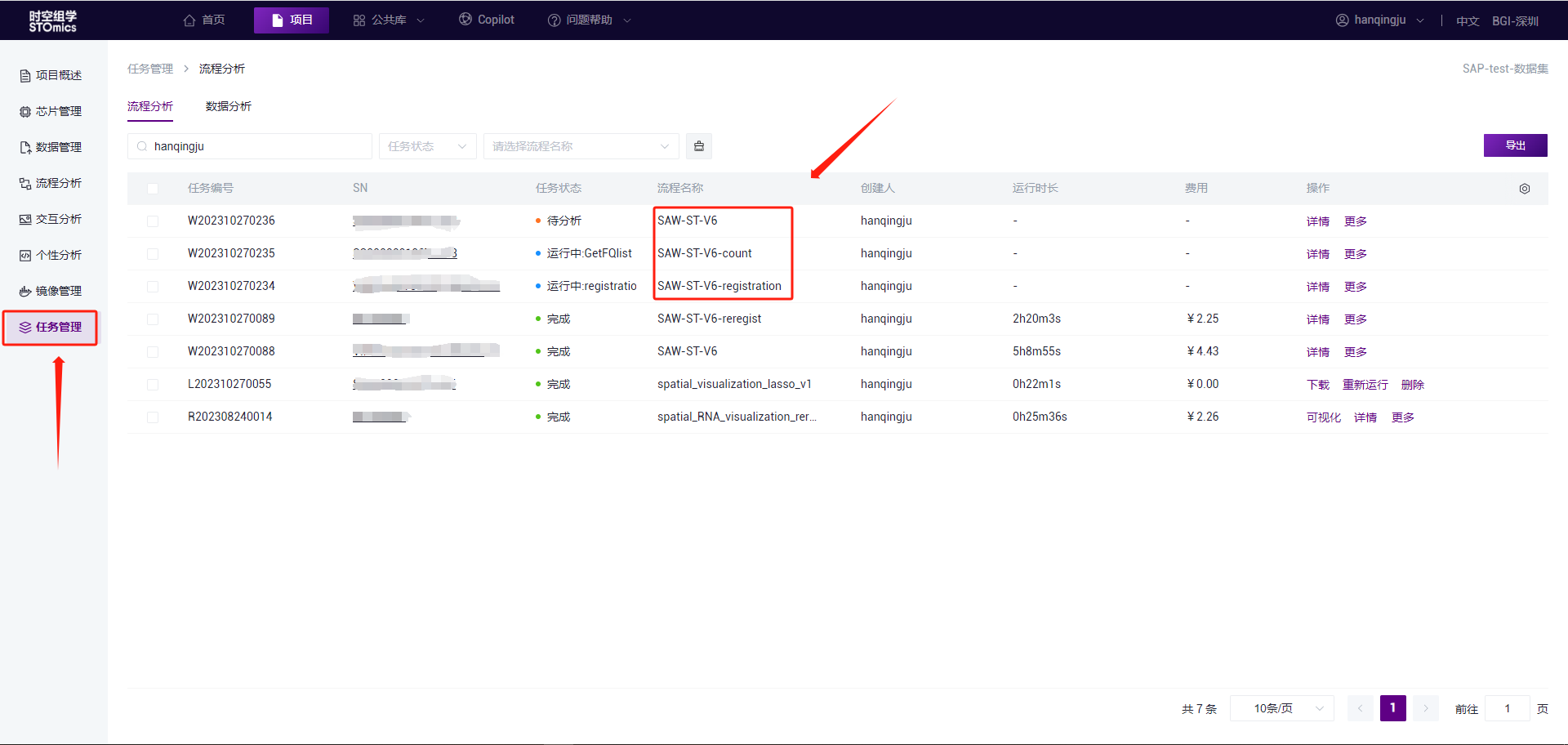
- 若是运行完 SAW-ST-V6/V7 的数据,手动操作后,点击“投递流程”按钮,弹框参数说明如下:

a. SAW-ST-V6/V7 流程:主要适用于 QC 成功的数据,手动操作完成后,可直接在该页面投递流程。

b. SAW-ST-V6/V7-count:主要适用于 QC 失败的数据,做完手动可以投递该流程。

c. SAW-ST-V6/V7-recut:主要适用于 QC 成功的数据,跑完全流程后,手动组织分割或者细胞分割,可以使用该流程。

d. SAW-ST-V6/V7-Registration:主要适用于 QC 成功的数据,做完手动拼接后可以使用该流程,参数自动填充。

如果是运行完比如: spatial_RNA_visualization_v5 或者patial_RNA_visualization_v6 等标准分析的任务,需要手动图像处理,手动完成后直接输出 IPR,用户自己在流程分析或者在芯片管理投递任务。具体操作参考流程分析。
在任务管理查看流程的状态。