可视化
可视化
访问
获取示例数据
本软件提供可供宾客访问的示例数据,通过登陆官网页面:https://www.stomics.tech/ -> 支持 -> Demo数据 -> Stereomap 访问。

数据管理
本系统提供可供用户访问的数据管理。通过STOmics Cloud系统:https://www.stomics.tech/sap/ ->项目管理 任意点击一个芯片号即可访问该芯片号相关任务列表。在任务列表中选择“任务状态”为“完成”的任务号即可访问数据。
可视化主页面
页面布局和功能:
- 系统参数区:从左至右依次分别为:分辨率、图像中心位置坐标、当前鼠标位置在图像坐标。
- 菜单栏区:Gen列表、图层名称列表、binsize列表、手动工具、聚类列表。
- 工具区:缩放进度条、图像操作(存储,读取,导出)。
- 主图区(主画布操作区):可视化基因表达空间分布以及对画布的操作。
- 图例区:展示基因/蛋白图例、聚类图例。


系统参数
Resolution:图像分辨率。
Center:图像中心坐标点。
Mouse position:鼠标在图像上的位置。
菜单栏区
菜单栏从左至右依次介绍。

- Gene/Protein:点击按钮即可展开Gene/Protein面板,二次点击为收起Gene/Protein面板。

- 图层选择:点击按钮即可展开图层下拉列表,二次点击为收起图层列表。图层选择列表展示所有可能的图像包含:Image图层(ssDNA/DAPI,CcellMask,TtissueMask)、IF蛋白图层、主分析图层(基因热图/蛋白表达图层,cell聚类图层)、UMAP图像,以及对系统组件进行控制。

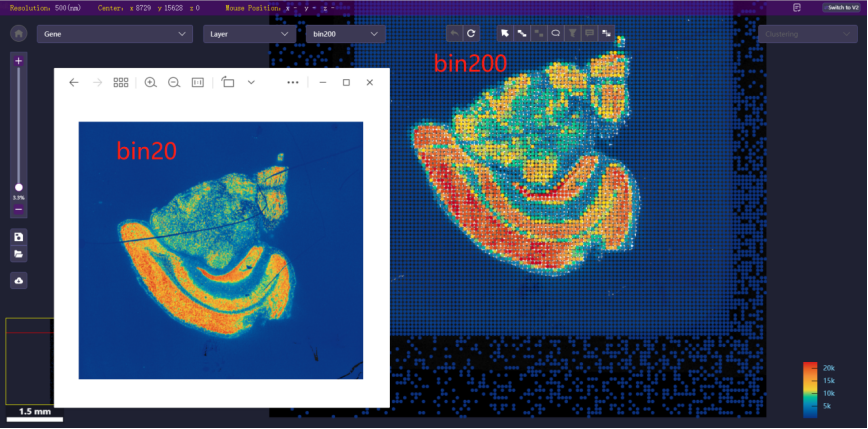
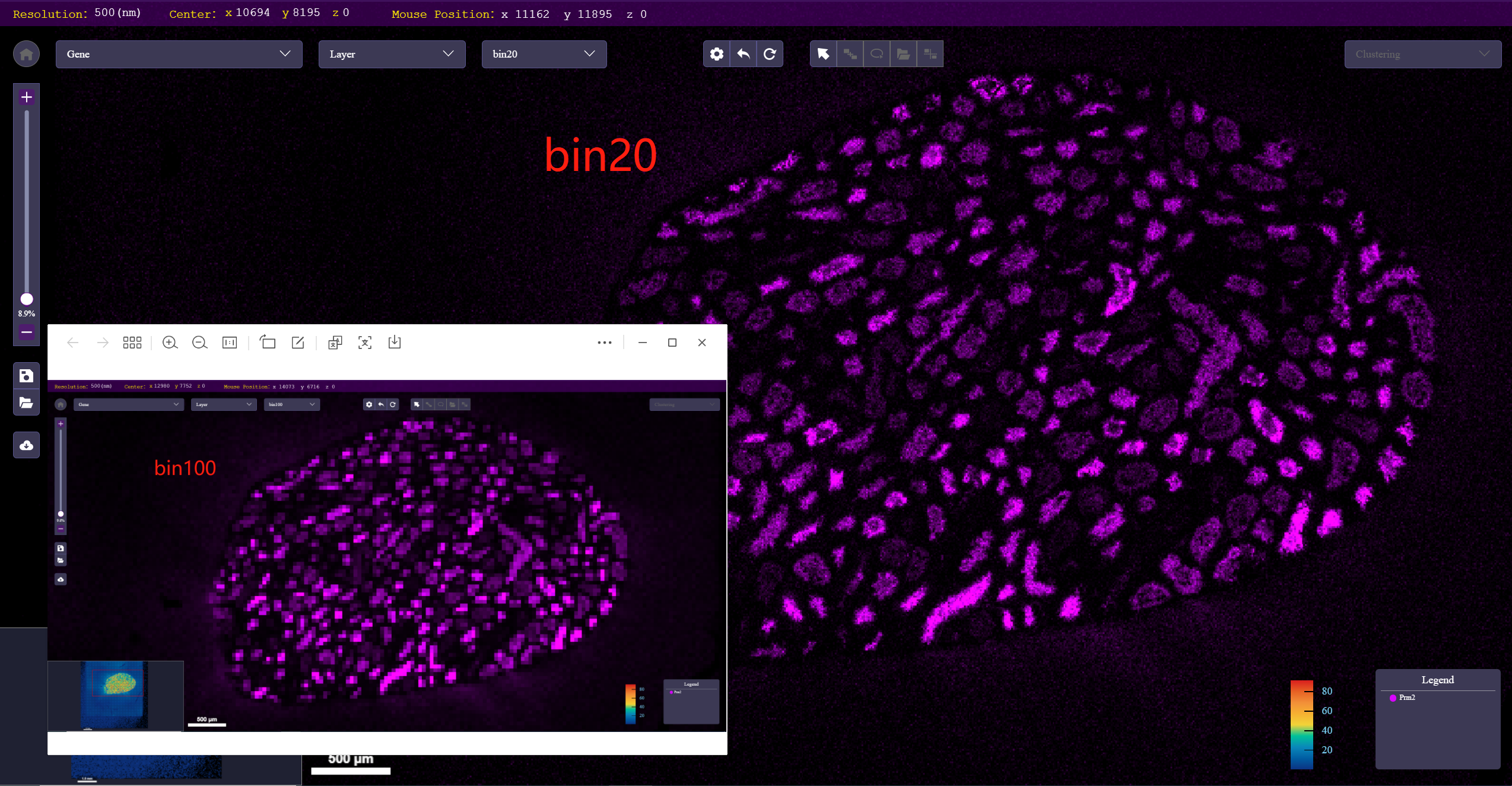
- binsize:展示当前芯片号的所有binsize,包括bin1、bin10、bin20、bin50、bin100、bin200、bin500以及cellbin(若该芯片号有cellbin数据则包含,否则侧不包含)。

- 设置按钮:包含图层附件工具的设置。

- 撤回按钮:点击按钮可撤回到上一步操作,最多支持10步。

- 重置刷新按钮:点击按钮可重置所有操作,恢复到初始状态。

- 游标按钮:点击即可退出手动工具模式。

- 手动配准按钮:点击即可进入手动配准模式,再次点击退出手动配准模式。
*手动配准工具教程详见手动配准。

- 套索按钮:点击即可进入套索模式,再次点击退出套索模式。
*套索工具教程详见章节套索。

- 过滤按钮:开发中。

- 标注按钮:开发中。

- 配准模板按钮:点击即可打开配准模板。
*配准模板工具教程详见配准模板。

- 聚类列表:展示聚类分析结果的列表。
Gene/Protein 列表
展示当前数据的基因/蛋白表达量列表。
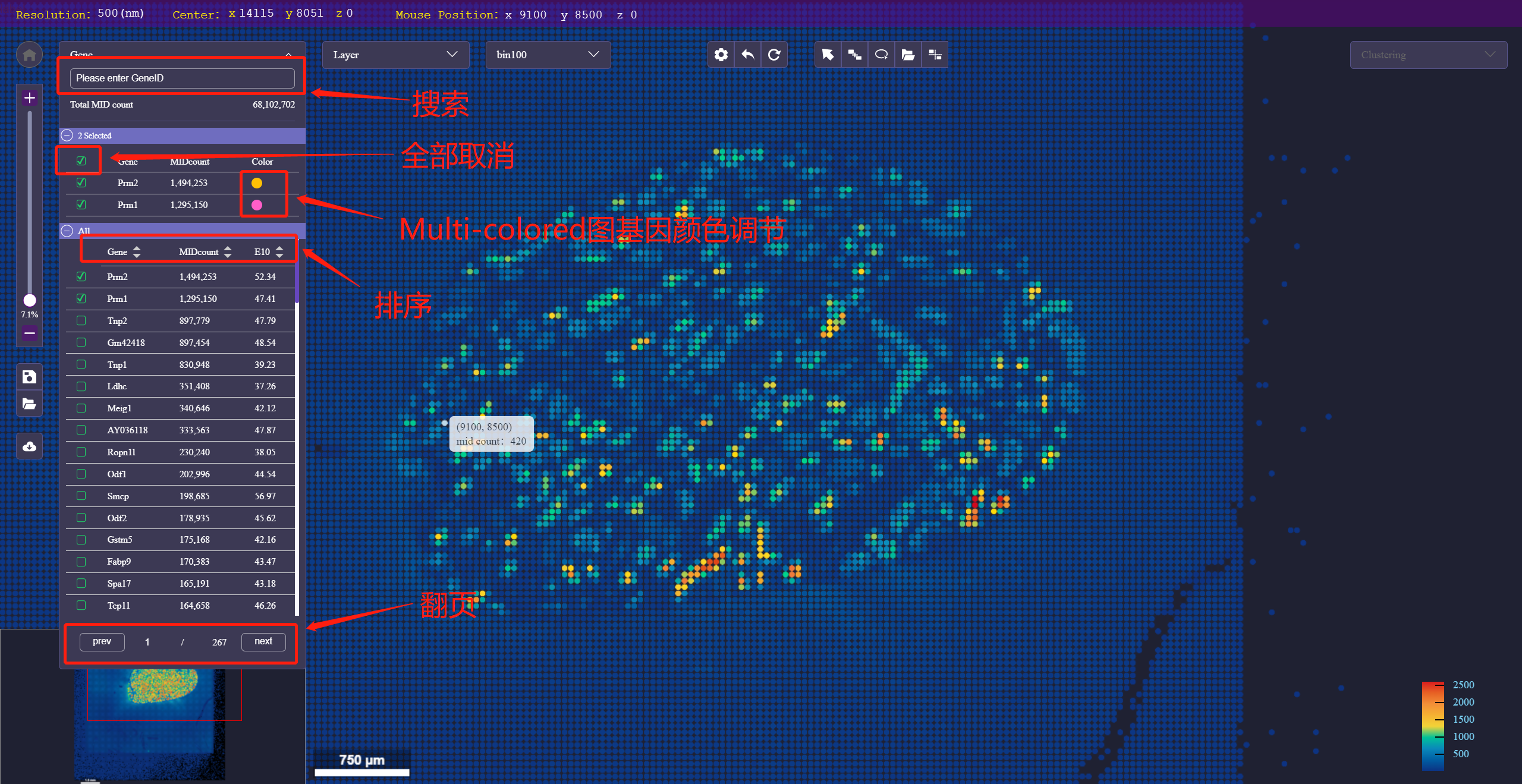
表头:“Gene”为基因名称、“MIDCount”为该基因的MID数、“E10”为基因的空间富集程度(E10)打分。
排序:单击表头右侧小箭头对表格进行升序或降序排列,默认按MID数降序进行排列。
筛选:在“Please enter GeneID…”(Please enter ProteinID…)处输入基因/蛋白名称,可以在表格中对基因进行搜索(不区分大小写,支持模糊、多名称搜索:使用英文逗号隔开)。
选择基因/蛋白:勾选复选框(基因名称前方的方框),会出现“Selected”分组,可选择使用Heatmap/multi-colored两种形式展示所选基因的表达丰度分布。教程详见章节2.4.3.1。
分页:点击上一页、下一页按钮进行翻页,在页码位置中输入数字按下回车即可跳转该页。
重置:点击”RESET”按钮重置到初始状态,恢复默认排序,清空已勾选项。
关闭:再次点击Gene/Protein 即可收起左侧Gene/Protein Table控制面板。
基因/蛋白merge图调色板
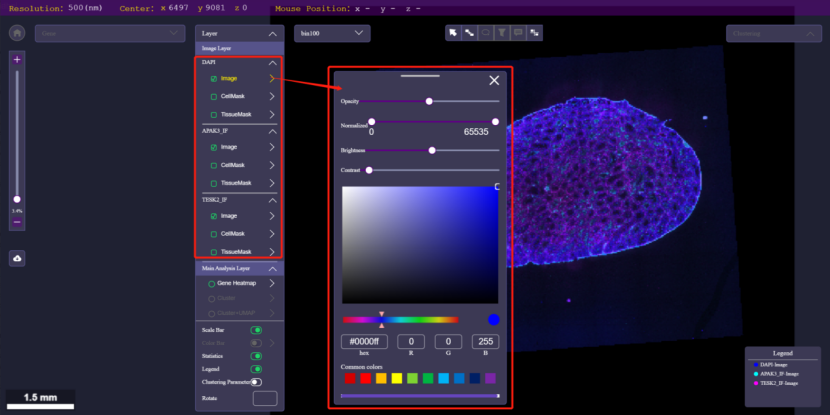
基因/蛋白merge图调色板包含以下五个参数:Opacity、Normalized、Brightness、Contrast、拾色器。如图2.4.2.2.1 - 2为基因/蛋白merge图调色板。教程详见章节2.4.3.1。
Opacity滑块:滑动滑块调整热图区域中的散点透明度。
Normalization滑块区:对底图图像数据进行归一化,左右滑动或输入数值可调整其最大最小值。归一化公式:

Brightness滑块:滑动滑块或输入数值调节基因/蛋白图层的亮度。
Contrast滑块:滑动滑块或输入数值调节基因/蛋白图层的对比度。
拾色器:展示该基因/蛋白图层颜色。拖动拾色器上的小圆圈或拖动彩虹带即可修改颜色,支持手动输入颜色代码。
*点击常用颜色中的颜色也可更改颜色。

图层选择
点击按钮即可展开图层下拉列表,二次点击为收起图层列表。图层选择列表展示所有可能的图像包含:Image图层(ssDNA/DAPI,IF,CellMask,TissueMask等)、主分析图层(基因热图/蛋白表达图层,cellbin聚类图层,UMAP图像),以及对系统组件进行控制。
1.点击图层名称或点击图层名称的展开箭头即可打开图层控制面板。
2.不同类型的图层控制面板中展示的内容有所不同。
3.图层名称前的勾选框:通过勾选/反勾选来展示/隐藏图像。
不同binsize下的主分析图层可能有所不同,如:
当读取的文件中包含.cellbin.gef格式文件,则分辨率为cellbin时,会展示Cluster图层;
当读取的文件中包含.spatial.cluster.h5ad格式文件,则分辨率为bin200时,会展示Cluster和Cluster + UMAP图层。
Image底图控制面板
点击图层名称即可调节颜色、透明度、归一化、亮度、对比度等参数,使用方法可参考2.4.2.2.1 -2 基因/蛋白多颜色图调色板。
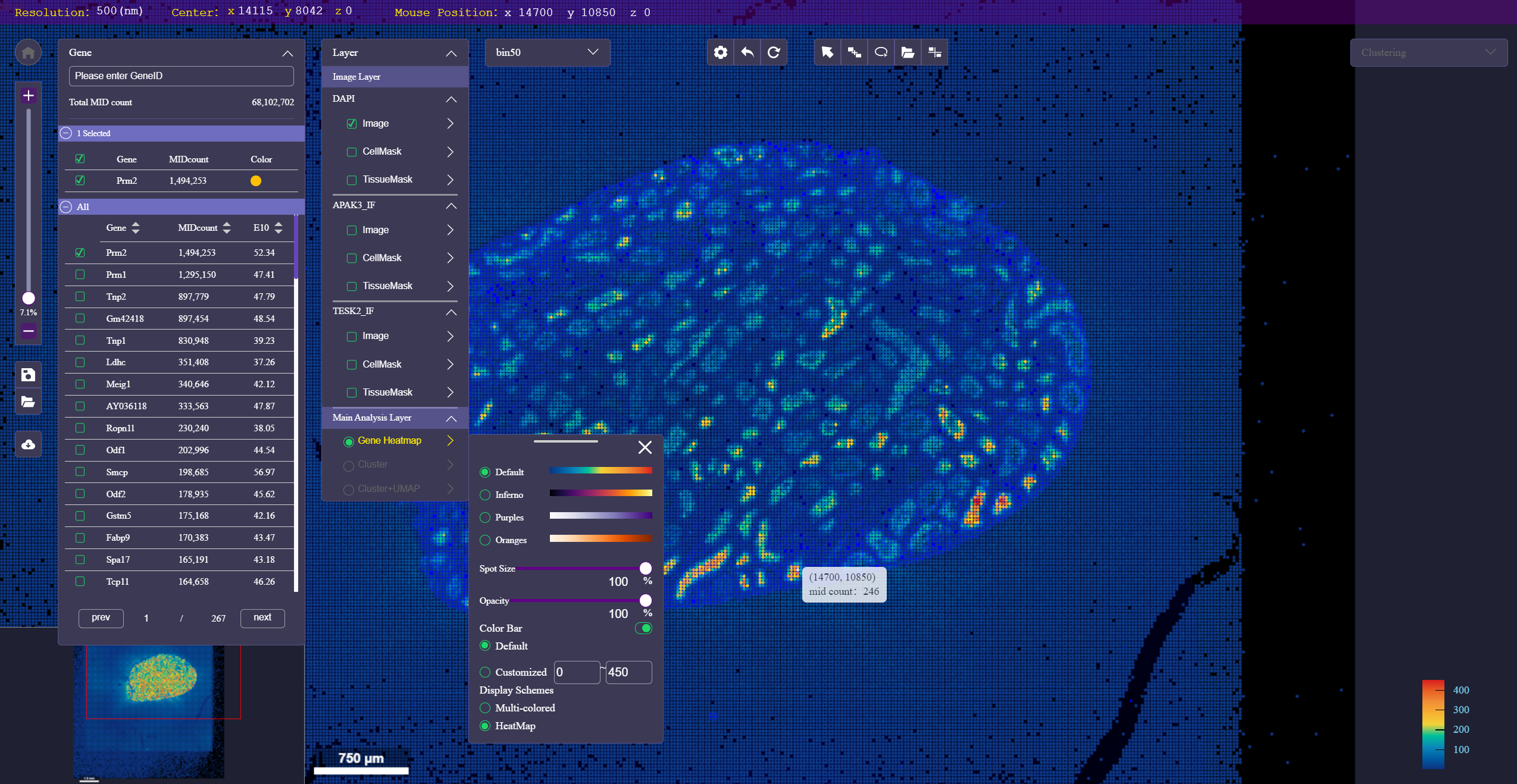
基因热图的控制面板
配色方案:提供四种热图配色,可自行选择。
散点大小(Spot Size):滑动滑块或者输入数字调整热图区域中的散点大小。
透明度(Opacity):滑动滑块或者输入数字调整热图区域中的散点透明度。
色阶尺(Color Bar):可控制开关、自动计算色阶尺范围或手动设置范围值。
展示形式(Display Schemes):仅在勾选基因/蛋白后显示此选项,用于选择单/多个基因/蛋白的展示形式。Heatmap即以热图形式展示;Multi-colored即以多颜色图形式展示,可在基因/蛋白面板中修改Selected分组内的各基因颜色。
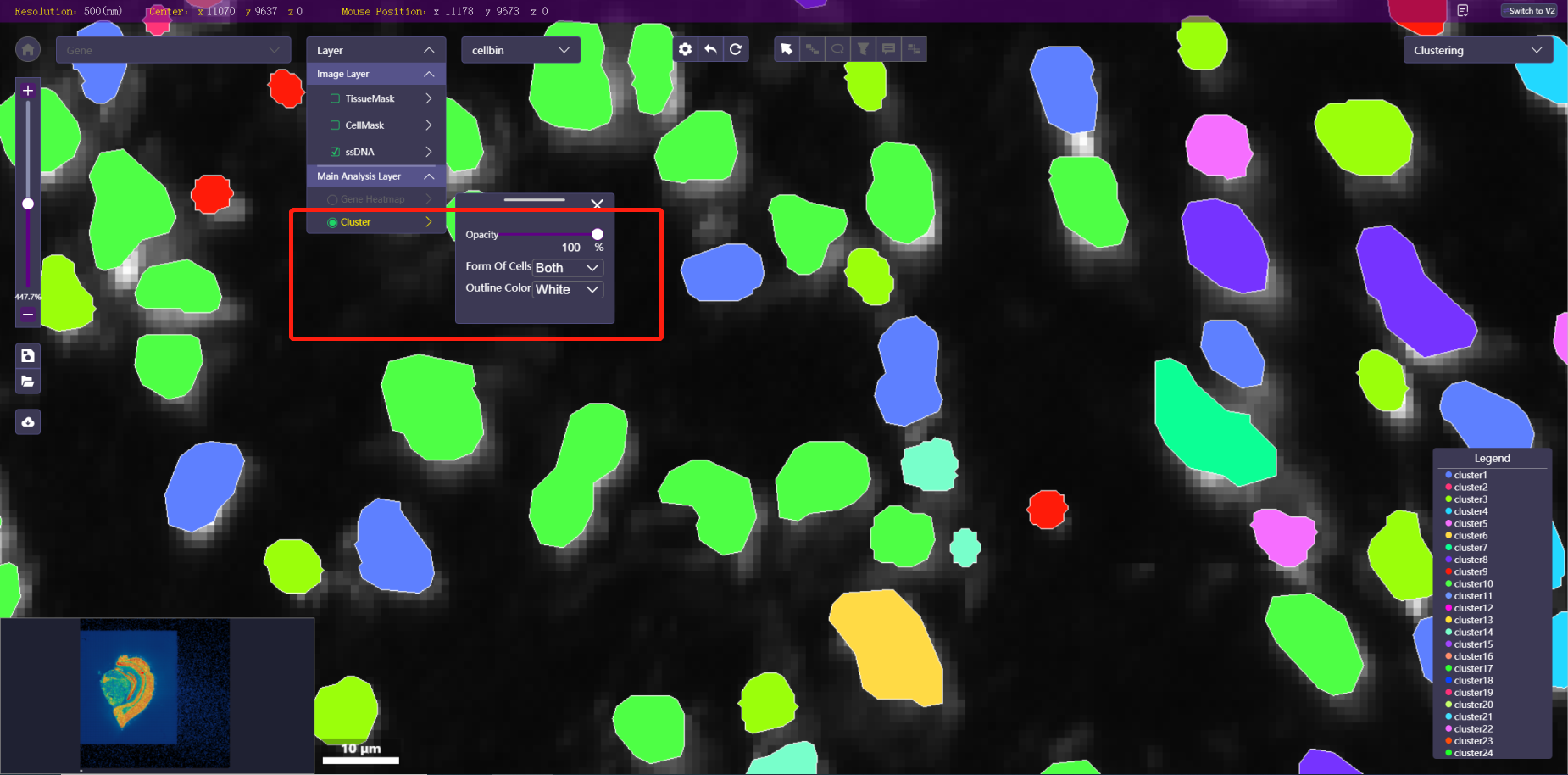
聚类图层控制面板
Opactiy:滑动滑块调整底图的透明度。
Form of Cells:包含Filled, Outlined, Both三个选项,可选展示方式为仅填充、仅轮廓或填充&轮廓。
Outlined Color:调节轮廓颜色,包含白、黑、绿及聚类颜色四种选项。
binsize选择
在基因/蛋白表达热图及聚类图层下,提供当前数据的不同分辨率的展示,包括bin1、bin10、bin20、bin50、bin100、bin200、bin500以及cellbin(若该芯片有cellbin数据则包含,否则不包含)。按住ctrl并滚动鼠标也可切换不同binsize。
*如果设置的 binsize 过小(如bin1),数据量过大可能会导致加载页面速度变慢,需要耐心等待,建议放大到局部后再切换较小的binsize。
手动工具菜单
设置

点击可打开设置面板,包含以下组件的开关及调整:比例尺、信息悬浮窗、图例、统计、缩略图、画布旋转。

撤回

点击图标可返回上一步操作,或使用快捷键Ctrl+Z。
重置

点击图标可清除当前所有操作,返回至任务打开时的初始状态。
游标

点击图标可退出手动工具模式。
手动配准

点击图标即可进入手动配准模式,再次点击可退出手动配准模式。
*手动配准教程详见手动配准。
套索

点击图标即可进入套索模式,再次点击退出套索模式。
*套索工具教程详见套索。
配准模板

点击图标即可打开表达矩阵的配准模板,可在此基础上对图像进行配准。
*配准模板教程详见配准模板。
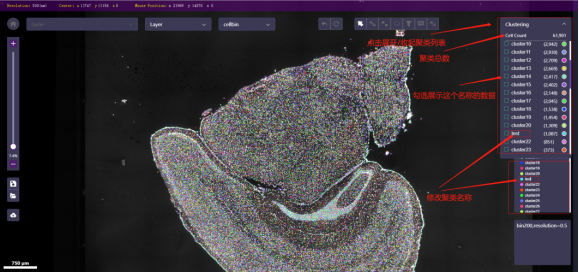
聚类列表
Clustering 页面展示聚类分析结果的列表。点击即可显示聚类列表,再次点击则关闭。
复选框:点击复选框,主图区展示选中的聚类数据。
名称:鼠标键入后可修改选中的聚类名称。
颜色:点击右侧打开调色板,拖动拾色器上的小圆圈/彩虹带即可修改选中的聚类颜色。
工具区
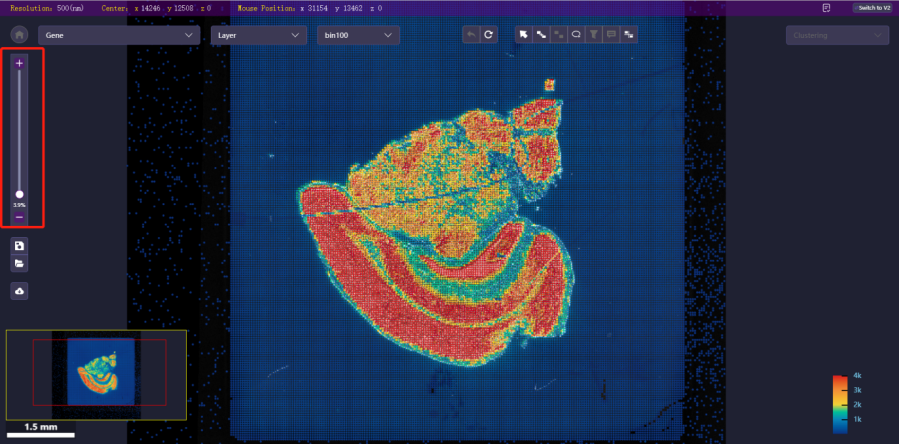
- 缩放进度条:控制主界面图像大小,拖动滑动条、滑动鼠标滚轮、或点击放大/缩小按钮均可对图像进行缩小或放大操作。如下图。
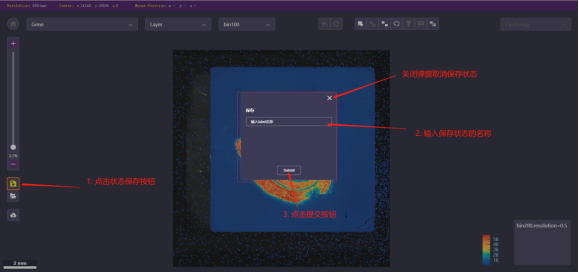
- 保存状态按钮:

保存当前图像的状态,便于后续可直接查看此次状态。
操作:点击按钮,打开保存状态弹窗,在弹窗中输入保存状态的名称,点击submit按钮提交即可保存。
*状态保存最多保存10条,超过10条后,每次保存都覆盖最早的文件。
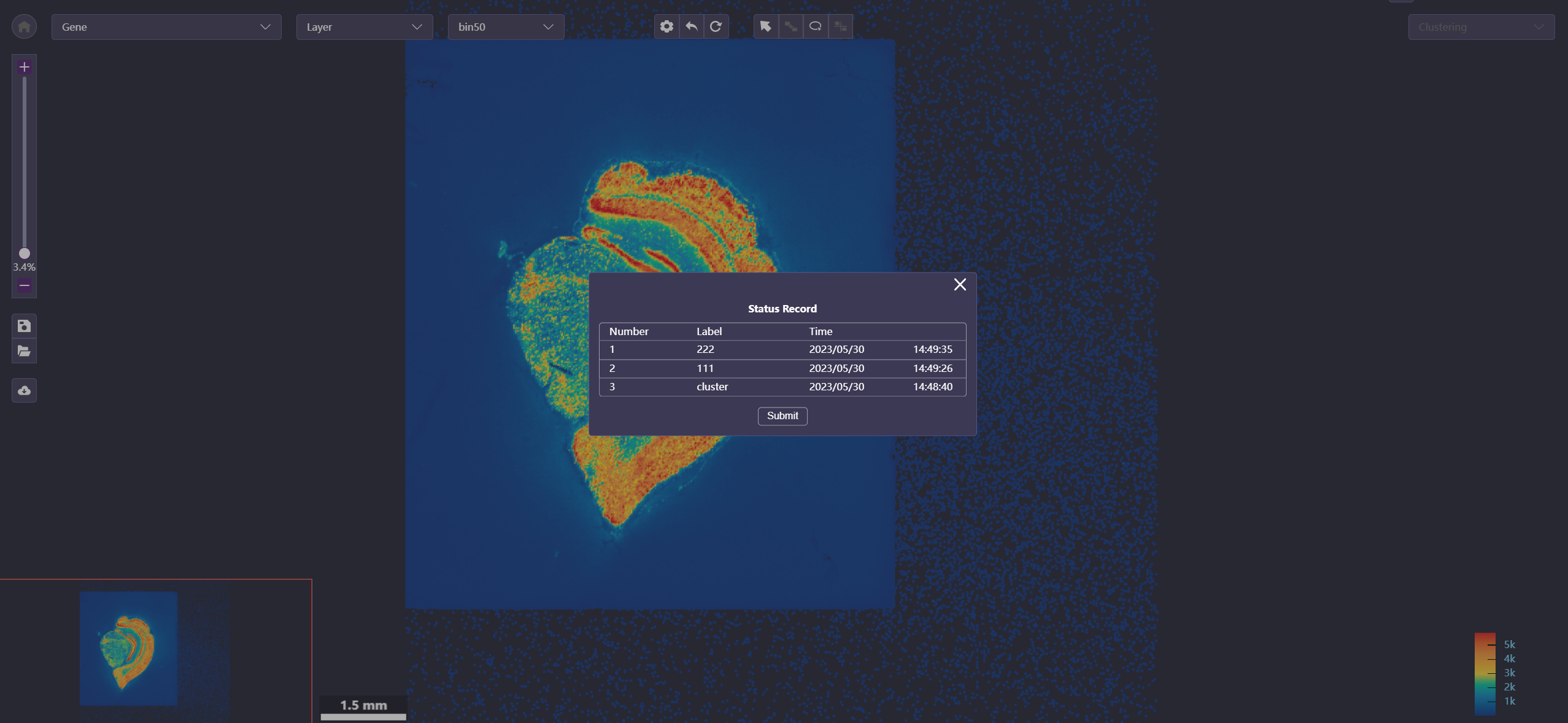
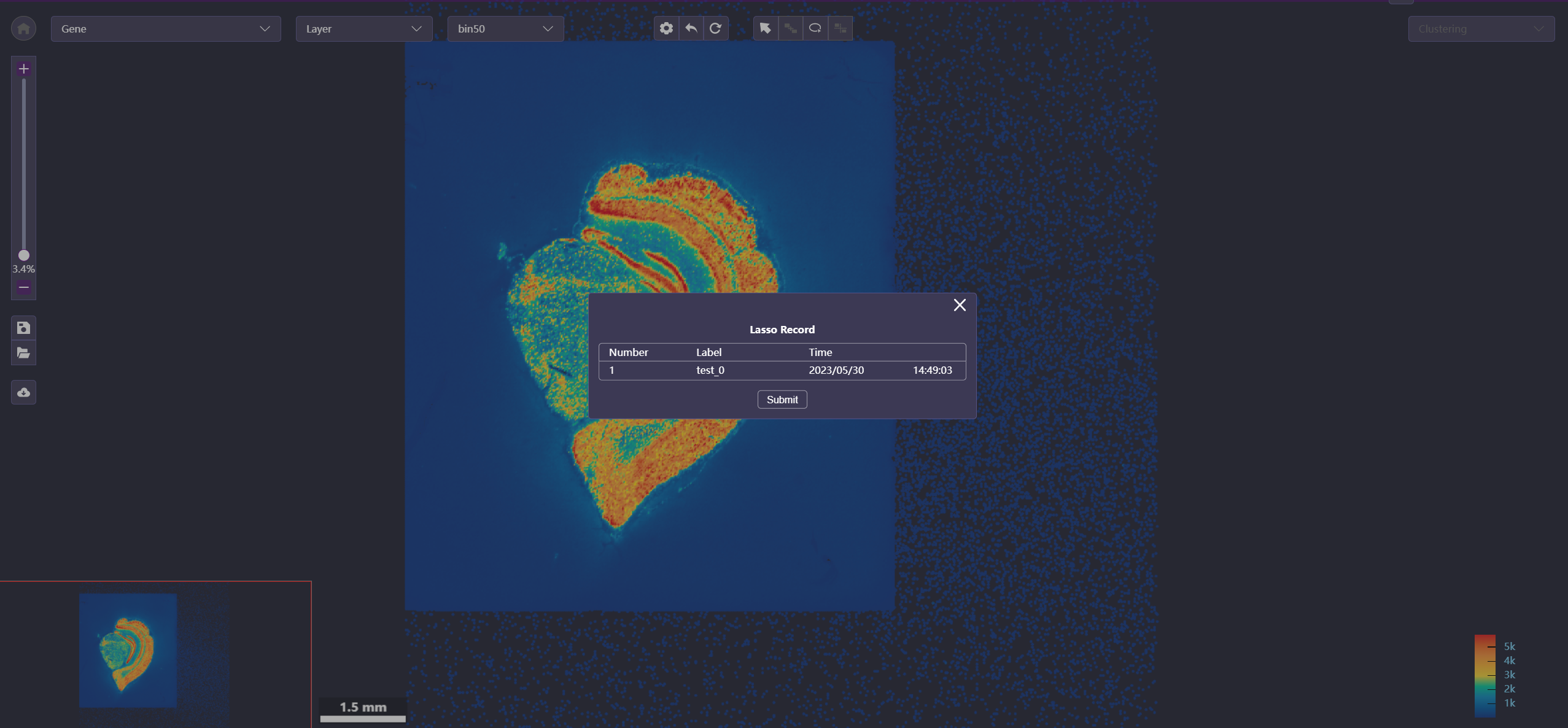
- 加载状态按钮:加载某次保存的状态,支持导入状态文件或套索文件。

操作:
加载状态文件时,点击“Load a Status File”,选择要加载的状态文件,点击确认即可打开选择的状态。
加载套索文件时,点击“Load a Lasso File”,选择要加载的套索文件,点击确认即可打开,继续进行套索区域的修改。
- 图像导出按钮:将当前画布上展示的图像保存至本地,可输入自定义文件名。

*Screenshot image:当前屏幕图像的截图。
*HD image:当前屏幕图像的高清图。若开启了legend图例,则图例会作为单独一张图片保存下来。

主图区
主画布区展示组织区域分布和表达量。
放大/缩小画布:滚轮向上/下滚动即可放大/缩小画布。
移动画布:使用鼠标拖拽(单机鼠标左键不放并移动鼠标)即可移动画布。
信息悬浮框:鼠标悬停在画布上会显示该点的坐标位置(x, y)和该点捕获到的基因种类数及对应的 MID 总数。可通过“设置”中的“Statistics”开关来控制悬浮框的显示与隐藏。

- 比例尺:画布左下方展示组织真实的比例尺。通过“设置”中的“Scale Bar”开关来控制比例尺的显示与隐藏。

- 色阶尺:画布右下方展示组织MID数与其对应的色阶尺。通过Gene Heatmap控制面板中的“Color Bar”开关来控制色阶尺的显示与隐藏、范围值设置。

- 切换binsize:使用键盘ctrl+滚轮向上/下可切换到前一个/后一个binsize。
例如binsize下拉菜单的顺序为:bin1,bin10,bin20,bin50,bin100,bin200,bin500,cellbin。当前binsize为bin100,操作ctr* + 滚轮向前(上)切换至bin50,操作ctr* + 滚轮向后(下)切换至bin200。
图例区
图例区包含两部分:上方为图例(基因/蛋白图例、聚类图例),下方为聚类参数。
基因/蛋白图例:在gene基因/蛋白列表中勾选复选框蛋白后,蛋白图例自动显示(通过图层选择中的"Legend"开关来控制图例的展示与隐藏)。
聚类图例:选择cellbin时,自动展示所有聚类,可通过聚类图例修改某个聚类label名称,也可通过聚类列表修改某个聚类label名称。(通过图层选择中的"Legend"开关来控制图例的展示与隐藏)。
聚类参数:当前聚类所用参数(通过图层选择中的"Clustering Parameter"开关来控制图例的展示与隐藏)。
操作演示(具体实现)
转录组数据可视化
勾选热图图层:在图层列表中选择Gene Heatmap图层。
展开基因列表:点击基因列表展开。
查看单/多基因Heatmap:在基因列表中点击基因名称前的复选框,即展示所选基因的热图,如下图。可通过右上角“…”按钮切换Heatmap和Multi-colored两种展示方式。

单/多基因勾选 Heatmap图层控制面板:当前图层以Heatmap形式展示时,可点击图层名称,打开控制面板,可调节热图颜色、散点大小、透明度以及色阶尺等。
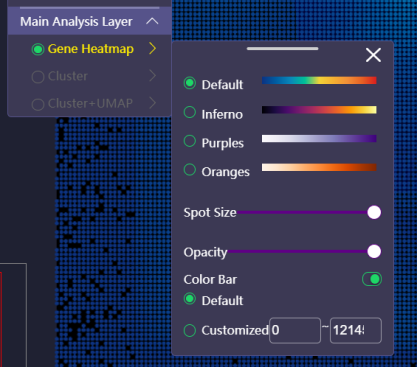
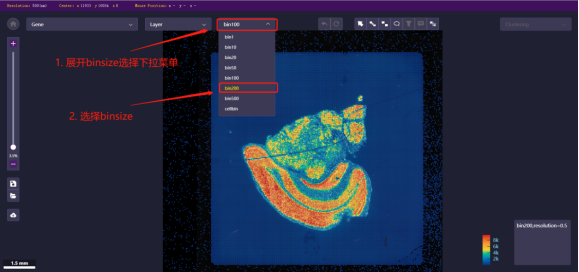
打开图层控制面板 切换binsize:展开binsize选择下拉菜单,选择bin即可。
*如果设置的 binsize 过小,可能会导致加载页面速度变慢,需要耐心等待,或放大到局部后在切换较小的binsize。
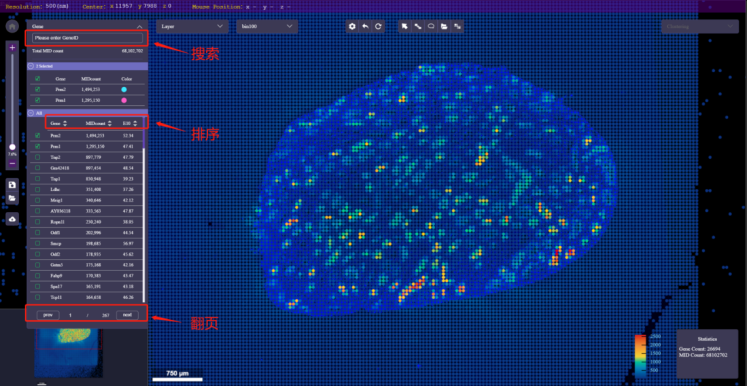
- 基因列表操作:
排序:单击表头右侧小箭头对表格进行升序或降序排列,默认按MID数降序进行排列。
搜索:在“Please enter GeneID…”(Please enter ProteinID…)处输入基因/蛋白名称,可以在表格中对基因进行搜索(不区分大小写,支持模糊、多名称搜索:使用英文逗号隔开)。
翻页:点击上一页、下一页按钮进行翻页,在页码位置中输入数字按下回车即可跳转该页。
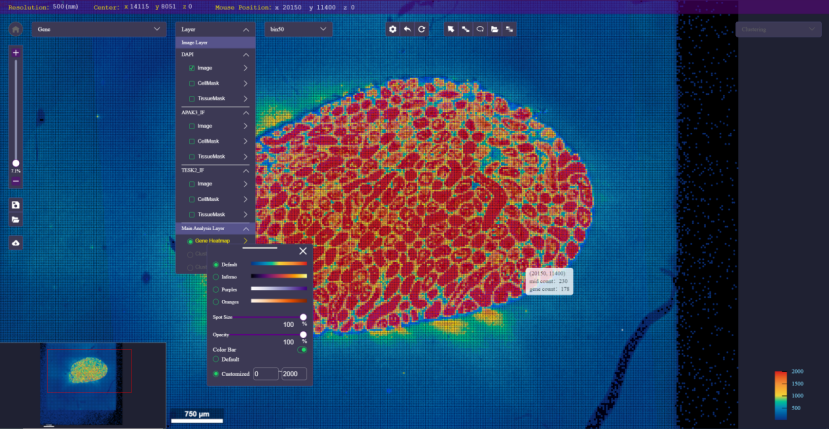
Color Bar自定义
在图层面板中,点击相应的Gene Heatmap图层名称打开控制面板,即可设置“Color Bar”,如下图。

- 色阶尺设置模式:
Default:自动计算模式。由软件自动计算得出的图像颜色。选择该模式则不能操作下方手动输入框。如图所示。
Customized:手动设置模式。点击后可在下方输入框中最小值和最大值。如图。
*手动设置模式下:
1.只能输入正整数。
2.最小值不大于最大值。
3.最大值的上线为50000000。
4.最大最小值数值不能相等。
5.该模式直至选择自动计算后结束。

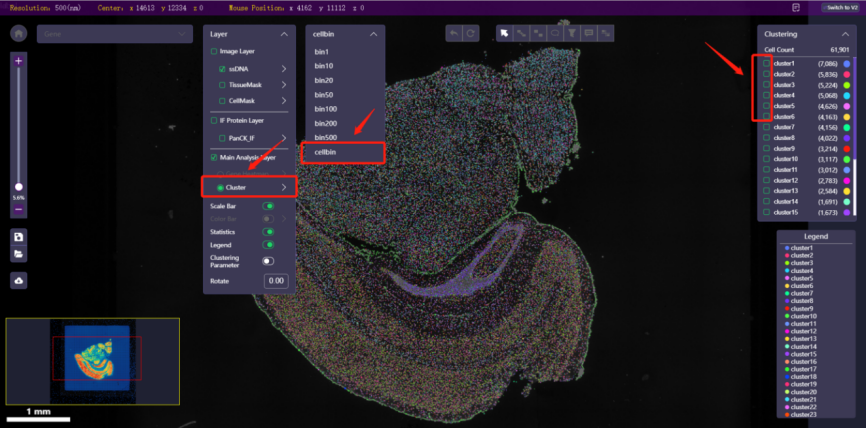
cellbin数据可视化
打开cellbin:点击菜单栏中的binsize选择下拉菜单选择cellbin;
打开cellbin图层控制面板:点击图层下拉菜单中的“Cel* Cluster”;
勾选聚类列表中相应的标签/label前的复选框;
退出cellbin:在binsize选择下拉菜单中选择普通bin即可。
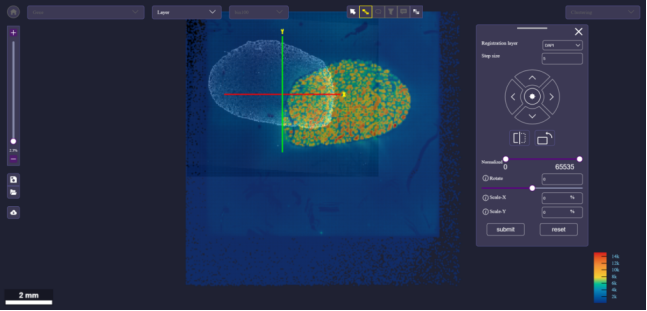
手动配准操作
点击手动配准工具按钮开启手动配准模式。
在"Registration layer"中选择需要配准的图像。
平移:在“Step size”中输入要位移的单位数量。本例中以500个单位平移,点击上/下/左/右方向按钮,每点击一次,图像向该方向平移500个单位。
特征点:X轴和Y轴的相交点为特征点,旋转及尺度缩放均以特征点为原点进行变换,特征点坐标保持不变。鼠标左键双击可改变特征点位置。

翻转:点击翻转按钮,图像绕Y轴镜像翻转。

- 90°旋转:点击此按钮,绕特征点逆时针旋转90度。

高精度配准:点击即可开启高精度配准,提交任务时由系统自动进行配准。再次点击取消高精度配准。
建议在拼接质量较高时使用此功能,否则请关闭此功能,手动配准至可接受的精度。

归一化(Normalized):对配准的图像数据进行归一化,左右滑动或输入数值可调整其最大最小值。
旋转(Rotate):围绕特征点对图像进行小角度旋转,可手动输入数字、点击上下箭头或拖动滑块调整角度,最小支持0.01°的调节。
X/Y轴尺度(Scale-X/Y):以特征点为中心,在X/Y轴方向对图像进行尺度放缩变换。可手动输入数字、点击上下箭头进行调节,最小调节尺度为图像在该方向长度的0.01%。
重置:点击“Reset”重置按钮,图像将重置到初始状态,即平移0个单位,翻转0次,旋转0°。
提交配准流程:点击“Submit”按钮提交配准流程。
退出手动配准:再次点击手动配准工具按钮即可退出,也可通过点击游标退出。
套索操作
进入套索模式:点击菜单栏中套索按钮进入套索模式,再次点击则退出。
新增套索区域:按住键盘ctr* + 鼠标左键并移动鼠标开始圈套。
删除套索区域:按住键盘alt + 鼠标左键并移动鼠标开始圈套。
*新增或删除区域都可进行多次操作。
完成一次圈套:点击Enter即可结束一次圈套,弹出套索统计窗口,分辨率为square bin(即bin1~bin500)时,统计窗口如图;分辨率为cellbin时,统计窗口如图。
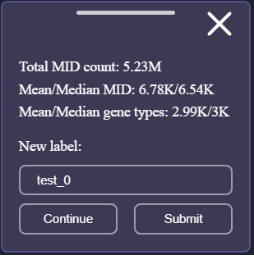
Square bin套索统计面板 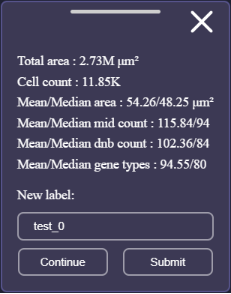
Cellbin套索统计面板 输入套索名称:在“New label”中填写套索区域名称,用户可根据套索区域的性质自定义名称。
修改圈套区域:点击Continue即可在已有的区域上继续修改,修改完成后再次点击Enter弹出窗口。
选择导出binsize:点击“Select bin size”下拉菜单,选择要导出的binsize即可,可支持多选。
删除所有套索图形:在弹出窗口中点击 “x”,关闭弹窗并删除所有套索图形。
提交套索任务:弹窗中点击“Submit”按钮即可提交任务,同时会保存该套索区域的记录,可通过“加载状态”工具打开,参见2.4.2.3节。
退出套索工具:再次点击套索按钮即可退出。(也可通过点击游标退出)
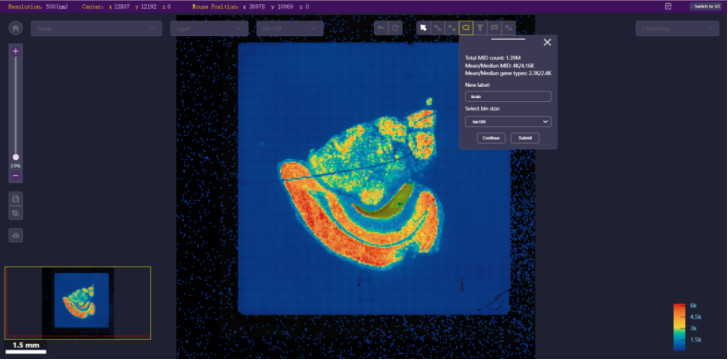
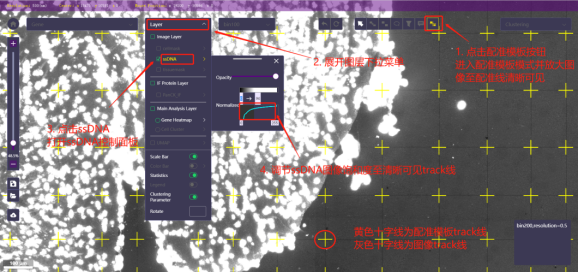
配准模板操作
打开配准模板:点击配准模板按钮打开配准模板(由多个黄色十字线组成)。
放大图像:在主图区域滑动鼠标滚轮即可放大图像。将图像放大至黄色十字线清晰可见即可。
调整图像对比度(以ssDNA为例):展开图层下拉菜单,点击ssDNA名称打开图层控制面板,调节Normalized使得ssDNA图像中可以看到清晰的track线即可,此时可以观察track线与配准模板的拟合效果。
退出配准模板模式:再次点击配准模板按钮即可退出。(也可通过点击游标退出)
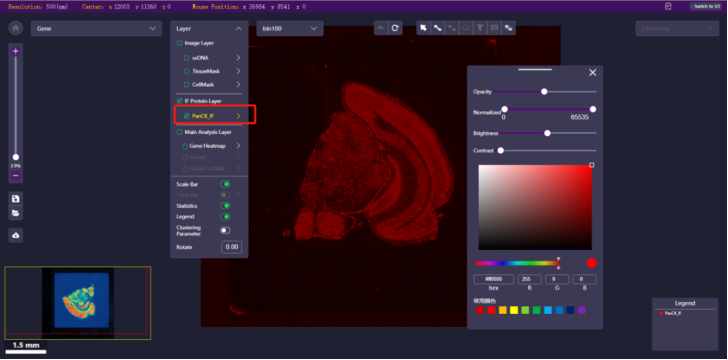
IF图层操作
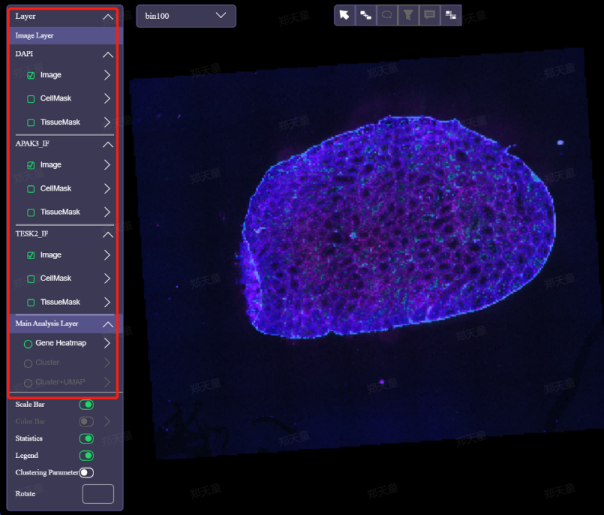
打开IF图层:在图层面板中勾选IF蛋白图像即可(Image对应原始图像,TissueMask对应该蛋白的组织分割区域,CellMask对应该蛋白的细胞分割区域)。蛋白名称会对应展示在图例中。如图42 。
调整蛋白颜色:不同的IF图层可以应用不同的颜色。点击图层名称打开图层控制面板,样式同图7 基因/蛋白多颜色图调色板。
关闭IF图层:取消勾选图层名称即可退出。